
3×3にパネルを並べて「臨兵闘者皆陳烈在前」を書く。

ここから二種類の表をつくる。
まずバイナリデータ表をつくる。
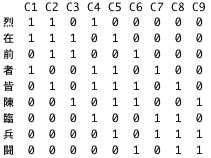
上記の表では、そのもの自身を含み隣接しているものについては「1」、それ以外は「0」を入れている。
数え上げやバイナリなどの離散データではないものも用意するため、「九字」相互の距離データに基づくクロス集計表も作成する。このとき、各文字間の距離をタイル中心からの距離とし、タテヨコ1文字分の距離を「1」として計算する。その他はこれに準じてナナメに1文字分は2の平方根(約1.41)等々とする。

CAで分析するときのスクリプトと実行結果は次の通り。
X<-read.table("bin_sample.txt", header=T)
#X<-read.csv("rvm_sample.csv", header=T)
test <- as.data.frame(X)
test.ca <- corresp(test, nf=9)
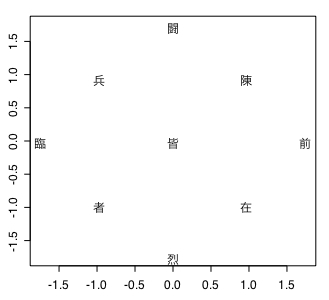
plot(test.ca$rs,type="n",ann=F)
text(test.ca$rs,rownames(test.ca$rs))
eigenvalues <- test.ca$cor^2
POV<- round(100*eigenvalues/sum(eigenvalues),2)
[1] 33.58 33.58 14.84 7.57 4.26 4.26 0.96 0.96 0.00
バイナリ表の分析結果では累積寄与率が67.16パーセントで低いにも関わらず、元のデータ構造を相当程度再現できている。
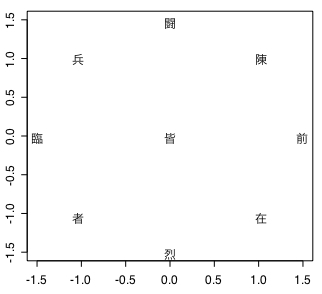
[1] 43.73 43.73 4.83 2.90 2.04 0.98 0.98 0.79 0.00
実測値表の結果はバイナリ表に比べて再現度が低いにもかかわらず、累積寄与率は87.46パーセントと高い値になっている。CAはPCAと比べて「データ構造の再現」では劣り「特徴別での分類」では勝ると言われているが、バイナリデータは後者の性質が有効に機能する。
PCAで使うスクリプトは以下の通り。
#X <- read.csv("rvm_sample.csv", header=T)
X <- read.table("bin_sample.txt", header=T)
test.pca <- prcomp(X)
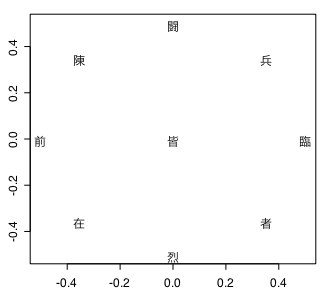
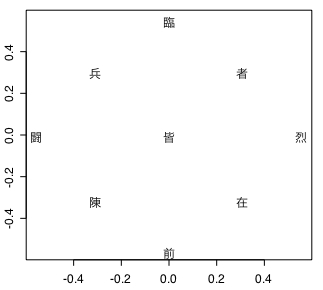
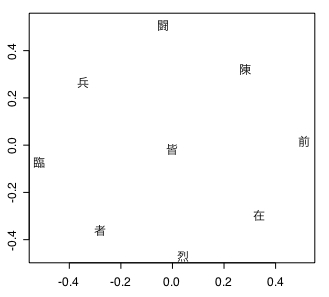
plot(test.pca$rotation, type="n", ann=F)
text(test.pca$rotation, rownames(X), col="black")
PCAPOV <- summary(test.pca)
Cumulative Proportion 0.305 0.610
バイナリ表の結果をみると累積寄与率は61.0パーセントしかなく、うまく再現できていない。
Cumulative Proportion 0.421 0.842
実測値表の結果は累積寄与率は84.2パーセントで高く、視認できた結果と合致している。CAとは逆にバイナリで結果が悪く、実測値表でよく再現できている。
バイナリ表も実測値表も対称データであるので、数量化IV類でも扱える。
X<-read.table("bin_sample.txt", header=T)
X<-read.csv("rvm_sample.csv", header=T)
#X.dist <- dist(X, method = "euclidean")
X.dist <- dist(X, method = "binary")
dissim <- isoMDS(X.dist, k=2)
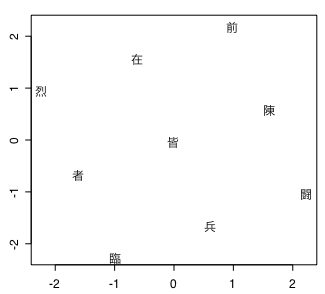
plot(dissim$points, type="n", ann=F)
text(dissim$points, rownames(X), col="black")
sdgm <- Shepard(X.dist, dissim$points, p=2)
STRESS <- dissim$stress
[1] 7.549709
データを距離化する関数 dist は色々な method が使える。通常は euclidean を使うが、バイナリデータを扱うので binary を使った。しかしCAの結果と比べるとあまり精度は高くない。stress値の数値も高すぎてどう判断してよいかわからない。
[1] 0.006496277
実測値に対しては stress値も十分に小さく、その通りの正確な結果となった。
CMDSのスクリプトは以下の通り。
X <- read.table("bin_sample.txt", header=T)
#X <- read.csv("rvm_sample.csv", header=T)
dissim <- cmdscale(X, eig=TRUE)
plot(dissim$points, type="n", ann=F)
text(dissim$points, rownames(X), col="black")
eigenvalues <- dissim$eig
MDSPOV<- round(100*eigenvalues/sum(eigenvalues),2)
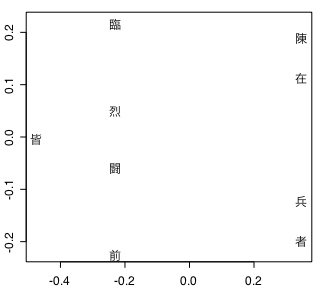
CMDSは「ユークリッド距離」データしか扱えないのでバイナリ表の分析はできない。メチャクチャな結果になっている。実測値表に関しては以下のように正確な図が描かれる。
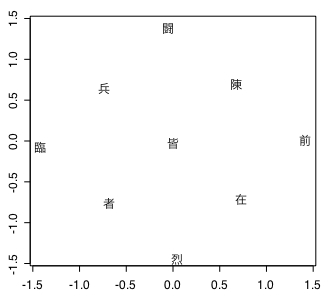
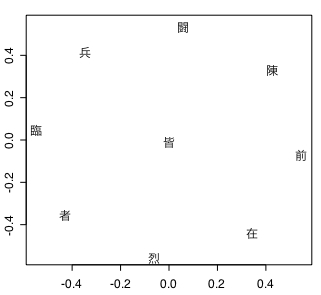
MDSはどちらも実測値データの再現性は高い。
CMDSはバイナリ表の値を「非類似度」的にするため「0」「1」を入れ替えると幾分まともな図になる。
NMDSでは結果の妥当性をシェパードダイアグラムで確認する。NMDS用スクリプトを実行したあと、下記のように入力すると図がかける。
plot(sdgm)
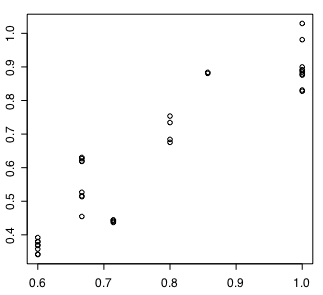
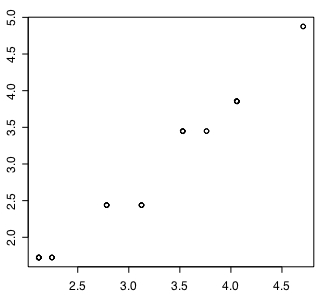
上がバイナリ、下が実測値表の場合である。
>> フリーソフトによるデータ解析・マイニング第26回「Rと対応分析」
>> フリーソフトによるデータ解析・マイニング第24回「Rと主成分分析」
>> フリーソフトによるデータ解析・マイニング第27回「Rと多次元尺度法」