
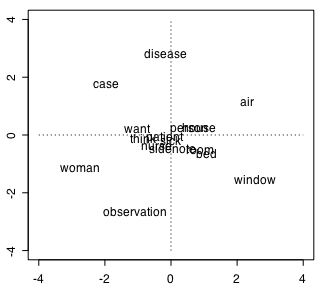
非類似度で表した hfv_items 同士の関係を2次元に縮約した結果は上図のように作図される。これを描く mds.R の中身は下記の通り。
X <- read.table("dissim.data", header=T)
dissim <- cmdscale(X)
plot(dissim, xlim=c(-4,4), ylim=c(-4,4), type="n")
segments(-4,0,4,0,lty=3)
segments(0,-4,0,4,lty=3)
text(dissim, rownames(X), col="black")
write.table(dissim, file="mdsout.data", sep="\t", row.names=T, col.names=F, quote=F)
xlim と ylim で描く図の範囲を指定している。segments は x=0 と y=0 の線を描く。分析結果の2次元座標は mdsout.data に書き出す。
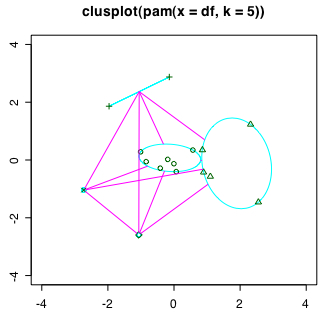
MDSの結果は上図のようにクラスタリングされる。この作図には cluster パッケージが必要となる。「パッケージインストーラ」を使って取得したあと「パッケージマネージャ」でロードしなくてはならない。tptbサンプル版ではこの図を描く部分はRファイルから削除してあるが下記のスクリプトを使って作図することは可能である。
clusplot(pam(df,5), xlim=c(-4,4), ylim=c(-4,4))
dfの中身は hclust.R を実行して mdsout.data から読み込むようになっているため、mds.R→hclust.Rの順で作業してから入力して作図する必要がある。
>> mds_pam.pdf
MDSの結果と pam によるクラスタリングを重ねてPDFファイルとして出力すると上記リンク先のファイルのようになる。
hclust.R の中身と結果は下記の通り。
Y <- read.table("mdsout.data")
df <- data.frame(Y, row.names=T)
hc <- hclust(dist(df), "ward")
plot(hc, hang=-1)
hc2 <- cutree(hc, k=5)
write.table(hc2, file="hc.data", sep="\t", row.names=T, col.names=F, quote=F)
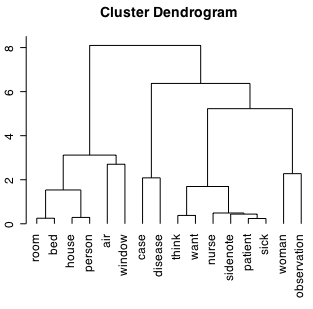
このデンドログラムは cutree 関数によって分割できる。分割結果はクラスターを表す数字が各項目に割り振られるだけなのでPerlスクリプトを使ってまとめる必要がある。上の例の場合、まとめると以下のようになる。
Cluster 1 (patient nurse sick sidenote think want )
Cluster 2 (room bed house person )
Cluster 3 (air window )
Cluster 4 (case disease )
Cluster 5 (woman observation )
この結果は pam 関数を使った先の例と若干異なる結果となっている。
pam は Partitioning Around Medoids の略で、デンドログラム作成に使った ward 法のような階層的クラスタリングとは異質な方法である。このため上のケースでは分割結果に違いが出た。pam や clara 等の cluster パッケージ関数については下記のサイトに説明がある。
>> 朱鷺の杜 Wiki
上で使ったデータでは pam と clara の結果に相違はなかった。ward 法と異なる部分の色を変えて分類すると下記のようになる。
Cluster 1 (patient nurse sick sidenote think want person)
Cluster 2 (room bed house air window)
Cluster 3 (observation )
Cluster 4 (case disease )
Cluster 5 (woman)
ward 法と pam 法の結果を比較してみた場合、語彙構造には確かに変化が見られる。このような変化は分析手法を変えた場合と同様、分析につかう hfv_items 数を増減しても当然生じてくる。語彙構造から推察できる意味構造自体もそれに伴って変化することは確実だが、問題はこの「変化」がどういう性質を持つのか、という点にある。
Copyright(c)2005-2012 ccoe@mac.com Allrights reserved.